(SARS-CoV-2-Immagine Credit Public Domain).
I ricercatori hanno sequenziato il genoma virale SARS-CoV-2 da campioni nello stato del Maharashtra e hanno trovato una combinazione unica di tre mutazioni che suggeriscono che il virus è in continua evoluzione per eludere la risposta immunitaria umana.
Con la pandemia COVID-19 che continua a diffondersi in diverse parti del mondo, SARS-CoV-2msi sta evolvendo per eludere il nostro sistema immunitario. Alla fine del 2020 nel Regno Unito, in Sud Africa e in Brasile sono state segnalate diverse nuove varianti, designate come varianti preoccupanti, che sembrano essere più contagiose del ceppo originale.
L’introduzione di nuove varianti e le loro conseguenze è importante per guidare la risposta della salute pubblica in un paese. Con l’enorme picco di COVID-19 osservato in India, specialmente nello stato del Maharashtra, il Governo ha aumentato il sequenziamento del genoma virale per identificare possibili nuovi mutanti e comprendere l’idoneità di nuovi ceppi. I ricercatori di diversi istituti indiani coinvolti nella ricerca SARS-CoV-2 hanno ottenuto campioni da viaggiatori internazionali nel Maharashtra. Circa il 5% dei campioni di sorveglianza è risultato positivo ed è stato sequenziato e l’intero genoma di SARS-CoV-2 è stato analizzato.
Il team ha anche analizzato la struttura cristallina delle prime 10 mutazioni della proteina spike del virus complessate con il recettore umano, l’enzima di conversione dell’angiotensina 2 (ACE2). I ricercatori hanno anche valutato l’effetto delle mutazioni sul legame a due anticorpi neutralizzanti utilizzando le strutture.
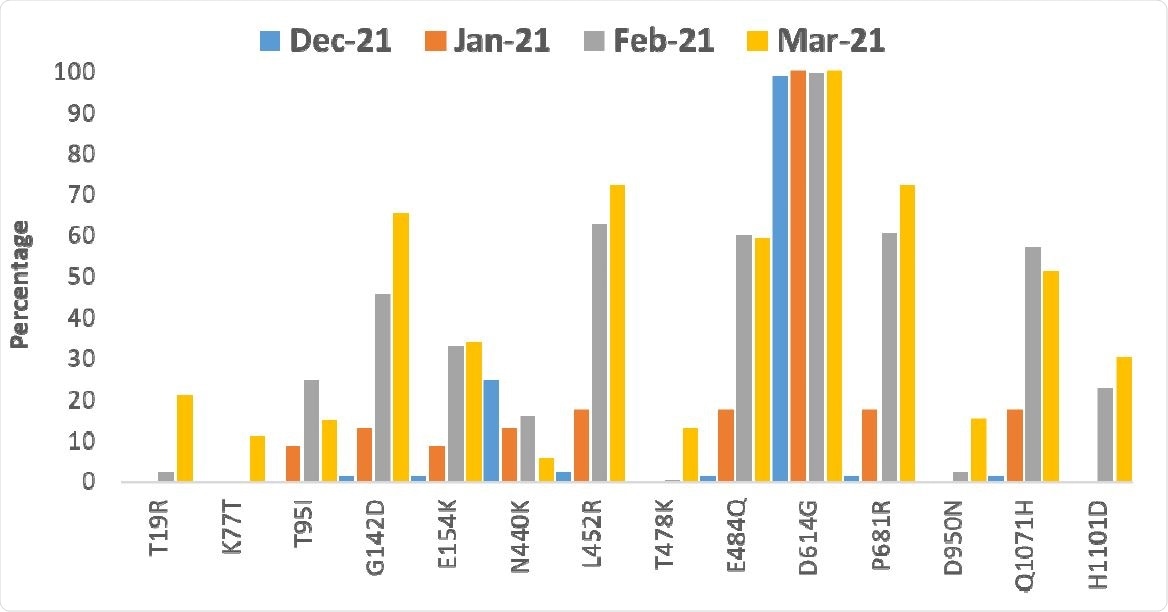
Identificazione delle mutazioni
Dopo aver analizzato 598 genomi virali interi, il team ha trovato 47 lignaggi G. Il più comune è stato il B.1.617, che è stato trovato in circa la metà dei genomi sequenziati. All’interno del clade B.1.617, i ricercatori hanno trovato quattro cluster collegati a specifiche mutazioni della proteina spike.
Hanno scoperto che le mutazioni L452R ed E484Q nel dominio di legame del recettore (RBD) della proteina spike e le mutazioni G142D e P681R al di fuori dell’RBD sono aumentate di frequenza dal gennaio 2021. Altre mutazioni includevano H1101D e T95I. Solo una piccola parte delle sequenze mostrava la variante B.1.1.7, in particolare a dicembre 2020.
Un gruppo di mutazioni non includeva E484Q e presentava mutazioni T19R e D950N nella proteina spike. Un’altra mutazione D111D si è verificata insieme alle mutazioni RBD L452R ed E484Q, ma non è stata osservata nel cluster senza la mutazione E484Q. L’analisi ha indicato un aumento della frequenza delle mutazioni non sinonime (quelle che modificano le sequenze proteiche) dal febbraio 2021.
Vedi anche:Come SARS-CoV-2 elude il sistema immunitario
Nel Maharashtra occidentale, luoghi come Mumbai, Pune, Thane e Nashik hanno mostrato diversi lignaggi oltre al dominante B.1.617 individuato nella parte orientale dello stato.
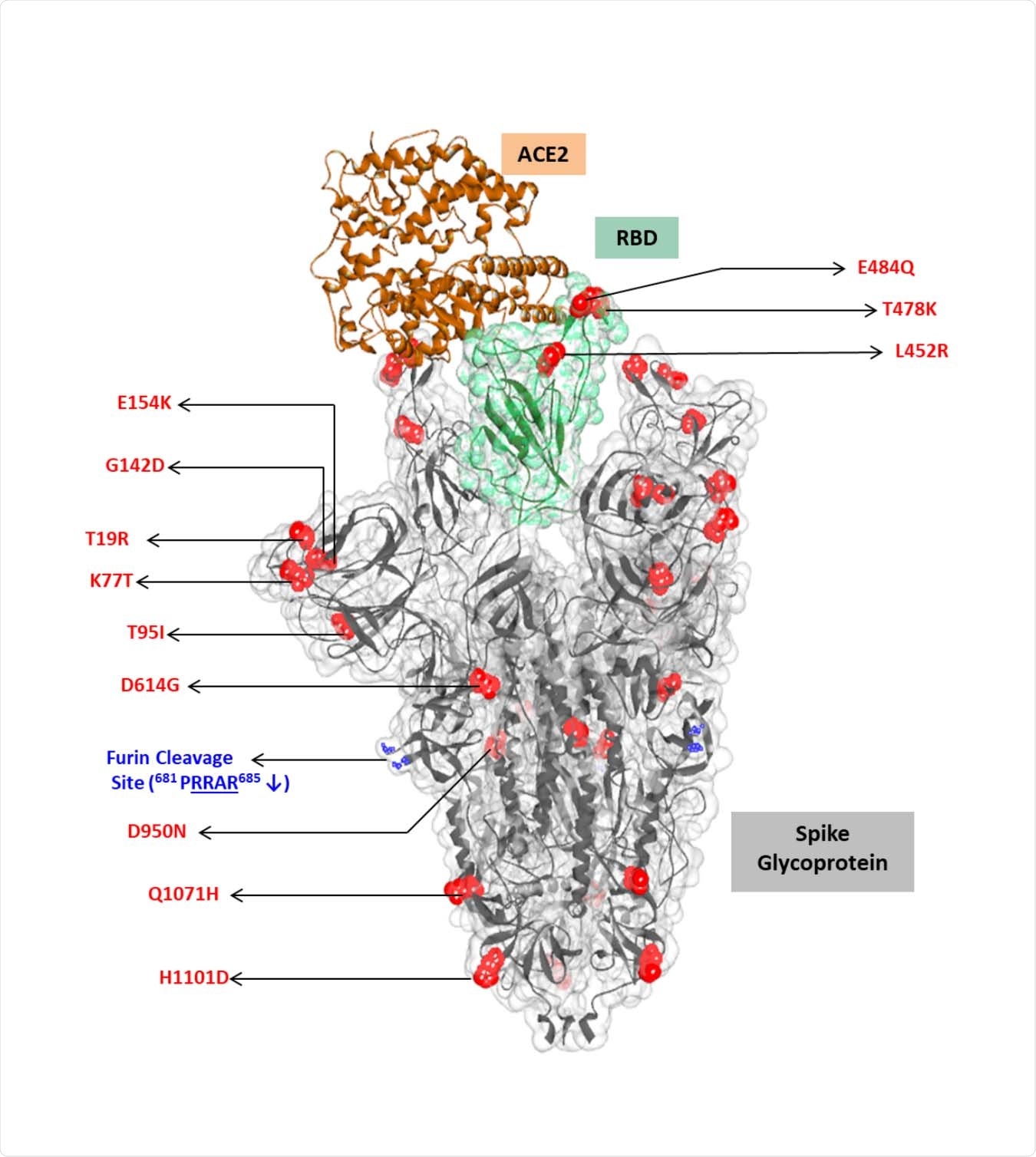
Le mutazioni modificano le interazioni con ACE2 e anticorpi
La struttura cristallina del complesso ACE2 con la proteina spike suggerisce che le mutazioni L452R ed E484Q riducono le interazioni intermolecolari e intramolecolari rispetto al virus wild-type. La mutazione L452R rimuove le interazioni idrofobiche con altri residui vicini. Tuttavia, il passaggio da idrofobo L452 a idrofilo 452R aumenta la stabilità del complesso, probabilmente a causa della sua maggiore interazione con le molecole d’acqua. La mutazione E484Q interrompe un legame elettrostatico nel RBD. La mutazione P681R nel sito di scissione della furina potrebbe aiutare una maggiore fusione della membrana e quindi causare una maggiore trasmissibilità, Entrambe le mutazioni L452R ed E484Q interrompono l’interazione degli anticorpi neutralizzanti REGN10933 e P2B-2F6 con la proteina spike, riducendo così il loro effetto di neutralizzazione.
Studi precedenti hanno dimostrato che la mutazione L452R riduce l’attività neutralizzante di diversi anticorpi monoclonali, compresi quelli in fase di sperimentazione clinica. Questa mutazione può anche sfuggire all’immunità cellulare, aumentare l’infettività virale e forse aumentare la replicazione virale. Pertanto, questa doppia combinazione mutante potrebbe sfuggire agli anticorpi monoclonali, richiedendo ulteriori studi.
Tripla mutazione
Mutazioni nei siti 452 e 484 sono state segnalate individualmente, ad esempio, nelle varianti B.1.427 e B.1.429 della California, che hanno la mutazione L452R. E484K è stato osservato nelle varianti segnalate da Regno Unito, Sud Africa e Brasile. Anche mutazioni nel sito P681 sono state osservate in alcuni lignaggi prima, così come la mutazione E484Q. La combinazione di tutte e tre le mutazioni qui riportate, L452R, E484Q e P681R, suggerisce che il virus sta evolvendo tratti simili in modo indipendente, adattandosi continuamente ai suoi ospiti umani.
L’aumento esponenziale dei casi del Maharashtra può essere collegato a questa tripla mutazione e ad altre mutazioni spike. Sebbene il lignaggio B.1.617 sia stato segnalato in molti altri paesi, la sua comparsa a livello locale necessita di un monitoraggio continuo per comprenderne l’impatto. I ricercatori concludono: “Lo studio ha esaminato le mutazioni della proteina S associate ai casi COVID19 nel Maharashtra osservate dal mese di febbraio 2021. Il continuo aumento della positività potrebbe essere attribuito a mutazioni distintive nella proteina spike e triple mutazioni funzionalmente significative. In base alle richieste GISAID, il lignaggio B.1.617 è stato segnalato da diversi paesi, tra cui Regno Unito, Stati Uniti, Svizzera, Germania, Singapore, ecc. Tuttavia, l’emergere di tali varianti locali durante la seconda ondata di COVID 19 in India deve essere ulteriormente indagato per il loro impatto sulla salute pubblica”.
Fonte: bioRxiv