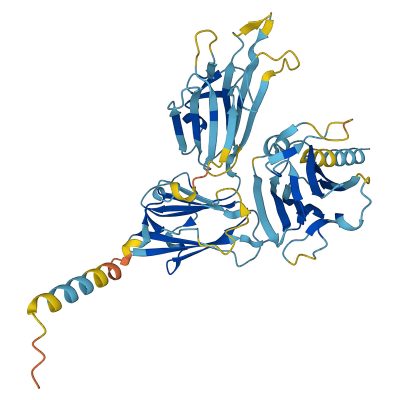
(Biologia digitale-Immagine: questa struttura prevista da AlphaFold rappresenta una proteina di superficie su uno stadio di un parassita della malaria che si sta sviluppando in un candidato al vaccino. DEEPMIND/EMBL-EB).
Dodici mesi fa, la società di intelligenza artificiale (AI) DeepMind ha sbalordito molti scienziati con il rilascio di strutture previste per circa 350.000 proteine, parte del lavoro riconosciuto come la svolta dell’anno 2021. Ieri, DeepMind e i suoi partner sono andati molto, molto oltre. L’azienda ha svelato le probabili strutture di quasi tutte le proteine conosciute, più di 200 milioni dai batteri all’uomo, un risultato sorprendente per l’IA e un potenziale tesoro per lo sviluppo di farmaci e gli studi evolutivi.
“Stiamo rilasciando ora le strutture dell’intero universo delle proteine”, ha affermato Demis Hassabis, fondatore e CEO di DeepMind, in una conferenza stampa a Londra.
L’individuazione e il rilascio strutturale delle proteine viene da AlphaFold, uno dei nuovi programmi di intelligenza artificiale che ha risolto il problema del ripiegamento delle proteine, la sfida di lunga data di derivare accuratamente le forme 3D delle proteine dalle loro sequenze di amminoacidi. Le strutture appena previste di AlphaFold sono state rilasciate ieri in un database esistente attraverso una partnership con l’Istituto europeo di bioinformatica (EMBL-EBI) del Laboratorio europeo di biologia molecolare. Il database “ha fornito ai biologi strutturali questo nuovo potente strumento in cui è possibile cercare la struttura 3D di una proteina quasi con la stessa facilità con cui è possibile eseguire una ricerca su Google per parola chiave”, ha affermato Hassabis.
Eric Topol, Direttore dello Scripps Research Translational Institute, ha fatto eco allo stupore di molti scienziati esterni. “AlphaFold è il singolare e epocale progresso nelle scienze della vita che dimostra il potere dell’IA”, ha twittato. “Con questa nuova aggiunta di strutture che illuminano quasi l’intero universo proteico, possiamo aspettarci che ogni giorno più misteri biologici vengano risolti”.
“Il rilascio della struttura di DeepMind è “notevole”, ha affermato Ewan Birney, vice Direttore generale di EMBL, durante la conferenza stampa. “Farà in modo che molti ricercatori in tutto il mondo pensino a quali esperimenti possono ora fare”.
Le proteine risolte da AlphaFold provengono da organismi che vanno dai batteri alle piante ai vertebrati, inclusi topi, pesci zebra e umani. Kathryn Tunyasuvunakool, una ricercatrice di DeepMind, ha affermato che AlphaFold ha impiegato circa 10-20 secondi per fare ogni previsione proteica. “L’azienda ha dovuto lavorare a stretto contatto con EMBL-EBI”, ha osservato, “per capire come presentare l’immenso numero di strutture nel database”.
Vedi anche:Cellule tumorali: l’AI le identifica
DeepMind afferma che più di 500.000 ricercatori hanno già utilizzato il database dal suo lancio lo scorso anno. Hassabis ha previsto una “nuova era nella biologia digitale” in cui gli sviluppatori di farmaci potrebbero passare dalle strutture di proteine previste dall’IA importanti per qualsiasi condizione medica, all’utilizzo dell’IA per progettare piccole molecole che influenzano quelle proteine e quindi curano una malattia.
Altri stanno usando le previsioni della struttura per sviluppare vaccini candidati, sondare questioni di biologia di base come come il cosiddetto complesso dei pori nucleari mantenga quali molecole entrano nel nucleo di una cellula o esaminare l’ evoluzione delle proteine quando la vita si è evoluta per la prima volta.
Hassabis, tuttavia, ha avvertito che il rilascio delle strutture è solo un punto di partenza. “Ovviamente c’è ancora molta biologia e molta chimica, che deve essere fatta”.