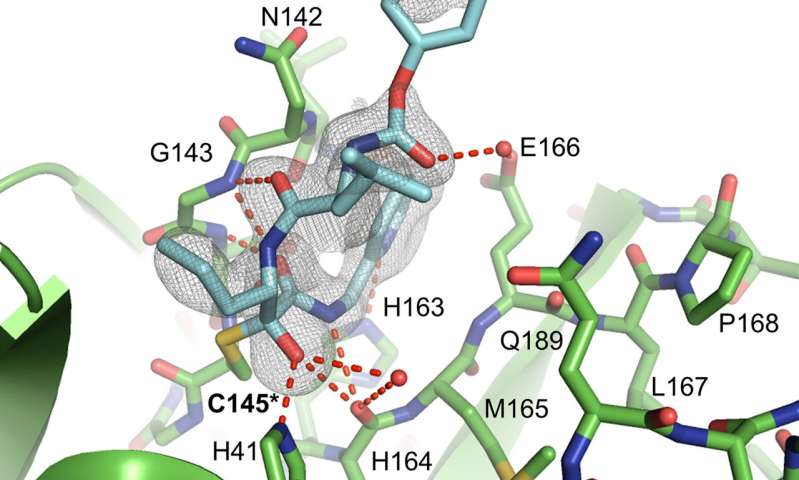
Immagine: struttura cristallina a raggi X della proteasi principale SARS-CoV-2 che interagisce con l’inibitore XII della calpaina (blu). È stato dimostrato che il composto antivirale adotta una configurazione atipica, chiamata posa di legame invertita, per adattarsi perfettamente al sito di legame proteico virale mirato. Credito: Michael Sacco della USF Health, utilizzando la cristallografia a raggi X.
SARS-CoV-2, il virus respiratorio che causa COVID-19, attacca il corpo in più passaggi. Entrare nelle cellule profonde all’interno dei polmoni e dirottare il meccanismo della cellula ospite umana per sfornare copie di se stesso: sono due dei primi passi, entrambi essenziali per l’infezione virale.
Un nuovo studio offre informazioni sulla progettazione di farmaci antivirali contro COVID-19 dimostrando che alcuni composti esistenti possono inibire sia la proteasi principale (M pro ), una proteina virale chiave richiesta per la replicazione di SARS-CoV-2 all’interno delle cellule umane, sia la proteasi lisosomiale catepsina L, una proteina umana importante per l’ingresso virale nelle cellule ospiti.
Lo studio, condotto dai ricercatori della University of South Florida Health (USF Health) Morsani College of Medicine e dell’Università dell’Arizona College of Pharmacy, è stato pubblicato oggi su Science Advances.
“Se riusciamo a sviluppare composti per arrestare o ridurre significativamente entrambi i processi – ingresso virale e replicazione virale – tale doppia inibizione può aumentare la potenza di questi composti nel trattamento dell’infezione da coronavirus”, ha detto il co-ricercatore principale dello studio Yu Chen, Ph.D ., Professore associato di medicina molecolare dell’USF Health. “Metaforicamente, è come uccidere due piccioni con una fava”.
I collaboratori della USF Health-University of Arizona (UA) si sono basati sul loro lavoro precedente, che ha identificato e analizzato diversi promettenti farmaci antivirali esistenti come candidati per il trattamento di COVID-19. Tutti i candidati erano stati scelti per perseguire l’obiettivo M pro per bloccare la replicazione di SARS-CoV-2 all’interno di cellule umane coltivate in laboratorio.
Due dei composti, gli inibitori della calpaina II e XII, non hanno mostrato la stessa attività contro M pro come un altro candidato farmaco chiamato GC-376 nei test biochimici. “Tuttavia, gli inibitori della calpaina, in particolare XII, hanno effettivamente funzionato meglio di GC-376 nell’uccidere SARS-CoV-2 nelle colture cellulari”, ha detto l’autore principale dello studio Michael Sacco, un dottorando nel laboratorio del Dr. Chen.
“Abbiamo pensato che se questi inibitori della calpaina fossero meno efficaci nell’inibire la proteasi principale del virus, avrebbero dovuto fare qualcos’altro per spiegare la loro attività antivirale“, ha detto Sacco. I ricercatori hanno appreso dalla ricerca condotta da altri gruppi, tra cui il collaboratore e co-ricercatore principale dello studio Jun Wang, Ph.D., di UA, che gli inibitori della calpaina possono bloccare altre proteasi, inclusa la catepsina L, una proteasi ospite umana critica coinvolta nella mediazione dell’ingresso di SARS-CoV-2 nelle cellule.

In questo ultimo studio, i ricercatori della USF Health hanno utilizzato tecniche avanzate, in particolare cristallografia a raggi X, per visualizzare come gli inibitori della calpaina II e XII interagivano con la proteina virale M pro. Hanno osservato che l’inibitore della calpaina II si adattava come previsto nei siti di legame mirati sulla superficie della proteasi principale SARS-CoV-2. Inaspettatamente, hanno anche scoperto che l’inibitore della calpaina XII ha adottato una configurazione unica, denominata “posa di legame invertita”, per adattarsi strettamente ai siti di legame proattivi M. (Una perfetta aderenza ottimizza l’interazione dell’inibitore con la proteina virale mirata, diminuendo l’attività enzimatica che aiuta la proliferazione di SARS-CoV-2).
Vedi anche:Scoperto uno dei trucchi più insidiosi del nuovo coronavirus SARS-CoV-2
“I nostri risultati forniscono utili informazioni strutturali su come possiamo progettare inibitori migliori per indirizzare questa proteina virale chiave in futuro”, ha detto il Dottor Chen.
“Oltre all’aumento della potenza (effetto farmacologico desiderato a una dose inferiore) per prendere di mira sia la proteasi virale M pro che la catepsina proteasica umana L, un altro vantaggio dei doppi inibitori è il loro potenziale di sopprimere la resistenza ai farmaci”, ha detto il Dottor Chen.
SARS-CoV-2 può mutare o modificare la sua sequenza genetica mirata. Queste mutazioni virali inducono la cellula umana a consentire al virus di attaccarsi alla membrana superficiale della cellula e inserire il suo materiale genetico e possono alterare la forma delle proteine virali e il modo in cui interagiscono con altre molecole (inclusi gli inibitori) all’interno della cellula.
Quando il virus muta in modo che possa continuare a riprodursi, può diventare resistente a un particolare inibitore, riducendo l’efficacia di quel composto. In altre parole, se la sequenza genetica del bersaglio virale (serratura) cambia, la chiave (inibitore) non si adatta più a quella specifica serratura. Ma supponiamo che la stessa chiave possa aprire due lucchetti per aiutare a prevenire l’infezione da COVID-19; in questo caso i due blocchi sono M pro , la proteina bersaglio virale e la catepsina L, la proteina bersaglio umana.
“È più difficile per il virus cambiare entrambe le serrature (due bersagli farmacologici) allo stesso tempo”, ha detto il Dottor Chen. “Quindi un doppio inibitore rende più difficile lo sviluppo della resistenza ai farmaci antivirali, perché anche se la proteina virale cambia, questo tipo di composto rimane efficace contro la proteina ospite umana che non è cambiata”.
I prossimi passi includeranno la risoluzione di come gli inibitori della calpaina interagiscono chimicamente e strutturalmente con la catepsina L.