Immagine: Public Domain.
La pandemia di COVID-19 è un duro richiamo al fatto che, sia in un singolo ospite umano che in un’ondata di infezione attraverso i continenti, la dinamica virale è spesso una storia di numeri.
In questo articolo, condotto da
- Department of Plant and Environmental Sciences, Weizmann Institute of Science, Israel
- Department of Molecular and Cell Biology, University of California, Berkeley, United States
- Department of Physics, Department of Applied Physics, and the Division of Biology and Biological Engineering, California Institute of Technology, United States
- Chan Zuckerberg Biohub, United States,
forniamo una fonte grafica unica e curata per i numeri chiave (basati principalmente sulla letteratura peer-reviewed) sul virus SARS-CoV-2 responsabile della pandemia. La discussione è incentrata su due grandi temi: i) la biologia del virus stesso; ii) le caratteristiche dell’infezione.
La pandemia COVID-19 ha chiarito brutalmente la necessità di ulteriori ricerche su molti aspetti dei virus. In questo articolo vengono raccolti dati sulle proprietà di base del virus SARS-CoV-2 e su come interagisce con il corpo (Figura 1). Discutiamo anche una serie di domande sul virus ed eseguiamo calcoli “back-of-the-envelope” per mostrare le intuizioni che possono essere acquisite conoscendo alcuni numeri chiave e usando il ragionamento quantitativo. È importante notare che permangono molte incertezze e, mentre i calcoli “back-of-the-envelope” possono migliorare la nostra intuizione attraverso controlli di integrità, non possono sostituire analisi epidemiologiche dettagliate.
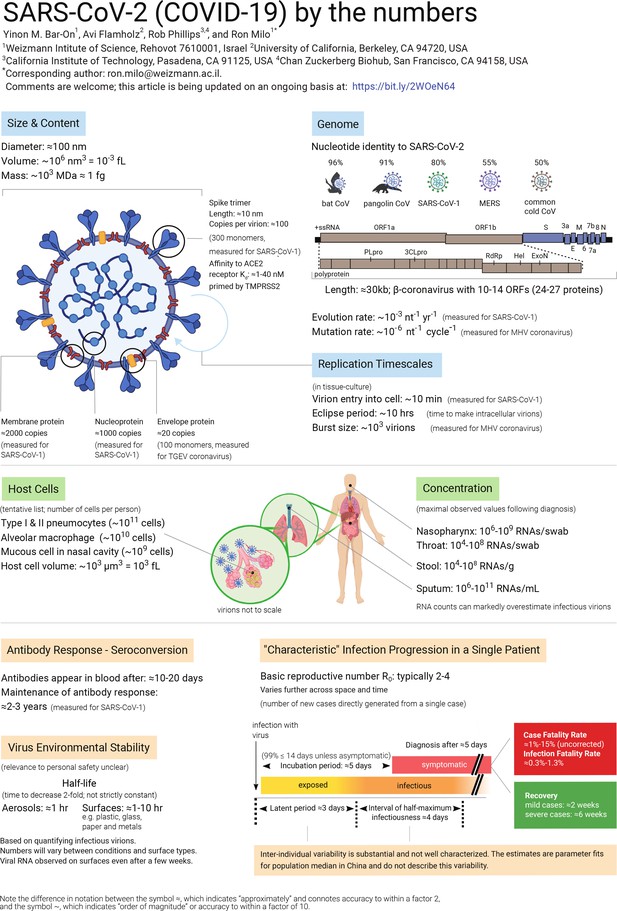
Immagine: il grafico mostra ciò che sappiamo sulle proprietà di base del virus SARS-CoV-2, come le sue dimensioni e il genoma, e su come interagisce con il corpo. Questi argomenti sono discussi ulteriormente nel testo, che include anche le fonti per tutti i valori elencati.
Sette domande su SARS-CoV-2
1. Quanto tempo impiega una singola persona infetta a produrre un milione di persone infette?
Se tutti continuassero a comportarsi come al solito, quanto tempo impiegherebbe la pandemia a diffondersi da una persona a un milione di vittime infette? Il numero di riproduzione di base, R0, suggerisce che ogni infezione genera direttamente altre 2-4 infezioni in assenza di contromisure come l’allontanamento fisico. Una volta che una persona è infetta, ci vuole un periodo di tempo noto come “periodo latente” prima che sia in grado di trasmettere il virus. L’attuale migliore stima del tempo di latenza mediana è di ≈3 giorni seguiti da ≈4 giorni di infettività prossima al massimo (Li et al., 2020a; He et al., 2020). Le durate esatte variano tra le persone e alcune sono contagiose per molto più tempo. Usando R0≈4, il numero di casi quadruplicherà ogni 7 giorni o raddoppierà ogni 3 giorni. Una crescita di 1000 volte (passando da un caso a 103) richiede 10 raddoppi da 210 ≈ 103; 3 giorni × 10 duplicati = 30 giorni, o circa un mese. Quindi prevediamo una crescita di 1000 volte in un mese, un milione di volte (106) in due mesi e un miliardo di volte (109) in tre mesi. Anche se questo calcolo è altamente semplificato, ignorando gli effetti di “super-spargitori”, immunità di gregge e test incompleti, sottolinea il fatto che i virus possono diffondersi a un ritmo sconcertante quando non vengono prese contromisure. Ciò spiega perché è fondamentale limitare la diffusione del virus mediante misure di distanziamento fisico.
2. Qual è l’effetto del distanziamento fisico?
Un esempio quantitativo altamente semplificato aiuta a chiarire la necessità di un distanziamento fisico. Supponiamo che tu sia infetto e incontri 50 persone nel corso di una giornata di lavoro, pendolarismo, socializzazione ed esecuzione di commissioni. Per arrotondare i numeri, supponiamo inoltre che tu abbia il 2% di probabilità di trasmettere il virus in ciascuno di questi incontri, in modo da infettare una persona nuova ogni giorno. Se sei contagioso per 4 giorni, in media ne infetti altri quattro, che si trovano all’estremità superiore dei valori di R0 per SARS-CoV-2 in assenza di distanziamento fisico. Se invece vedi cinque persone ogni giorno (preferibilmente meno) a causa del distanziamento fisico, infetterai 0,1 persone al giorno o 0,4 persone prima di diventare meno infettivo. L’effetto desiderato del distanziamento fisico è di far sì che ogni infezione attuale produca <1 nuove infezioni. Un numero di riproduzione (Re) inferiore a uno garantirà che il numero di infezioni eventualmente diminuirà. È di fondamentale importanza ottenere rapidamente Re <1, che è sostanzialmente più realizzabile che spingere Re a quasi zero attraverso misure di sanità pubblica.
Vedi anche: Due metri di distanza non sufficienti per fermare la trasmissione di SARS-CoV-2 con venti leggeri
3. Perché il periodo di quarantena iniziale è stato di due settimane?
Il periodo di tempo dall’infezione ai sintomi è definito periodo di incubazione. Il periodo mediano di incubazione SARS-CoV-2 è stimato in circa 5 giorni (Lauer et al., 2020). Eppure c’è molta variazione da persona a persona. Circa il 99% di quelli che mostrano sintomi li mostrerà prima del giorno 14, il che spiega il periodo di confinamento di due settimane. È importante sottolineare che questa analisi trascura le persone infette che non mostrano mai sintomi. Dal momento che le persone asintomatiche non vengono di solito testate, non è ancora chiaro quanti casi simili ci siano o per quanto tempo le persone asintomatiche rimangono infettive.
4. Quanto è simile SARS-CoV-2 ai comuni virus del raffreddore e dell’influenza?
SARS-CoV-2 è un beta-coronavirus il cui genoma è un singolo filamento di RNA ≈30 kb. L’influenza è causata da una famiglia di virus RNA completamente diversa chiamata virus influenzale. I virus dell’influenza hanno genomi più piccoli (≈14 kb) codificati in otto distinti filamenti di RNA e infettano le cellule umane in modo diverso rispetto ai coronavirus. Il “raffreddore comune” è causato da una varietà di virus, tra cui alcuni coronavirus e rinovirus. I coronavirus che causano il raffreddore (ad es. Ceppi OC43 e 229E) sono abbastanza simili alla SARS-CoV-2 in lunghezza del genoma (entro il 10%) e al contenuto genico, ma diversi dalla SARS-CoV-2 in sequenza (identità nucleotidica ≈50%) e gravità da infezione. Un aspetto interessante dei coronavirus è che hanno il più grande genoma di qualsiasi virus RNA noto (≈30 kb). Questi grandi genomi hanno indotto i ricercatori a sospettare la presenza di un “meccanismo di correzione di bozze” per ridurre il tasso di mutazione e stabilizzare il genoma. In effetti, i coronavirus hanno una esonucleasi di correzione delle bozze chiamata ExoN, che spiega i loro bassi tassi di mutazione (~ 10–6 per sito per ciclo) rispetto all’influenza (≈3 × 10–5 per sito per ciclo; Sanjuán et al., 2010). Questo tasso di mutazione relativamente basso sarà di interesse per studi futuri che prevedono la velocità con cui i coronavirus possono eludere i nostri sforzi di immunizzazione.
5. Quanto si sa sul genoma e sul proteoma SARS-CoV-2?
SARS-CoV-2 ha un genoma di RNA a senso positivo a singolo filamento che codifica per 10 geni alla fine producendo 26 proteine secondo un’annotazione NCBI (NC_045512). Com’è che 10 geni codificano per> 20 proteine? Un gene lungo, orf1ab, codifica una polipoteina che viene suddivisa in 16 proteine da proteasi che sono esse stesse parte della polipoteina. Oltre alle proteasi, la polipoteina codifica un RNA polimerasi e fattori associati per copiare il genoma, una esonucleasi di correzione di bozze e molte altre proteine non strutturali. I restanti geni codificano prevalentemente per componenti strutturali del virus: i) la proteina spike che lega il recettore cognato su una cellula umana o animale; ii) una nucleoproteina che impacchetta il genoma; iii) due proteine legate alla membrana. Sebbene gran parte del lavoro attuale sia incentrato sulla comprensione del ruolo delle proteine ”accessorie” nel ciclo di vita virale, stimiamo che attualmente sia possibile attribuire chiare funzioni biochimiche o strutturali a circa metà dei prodotti del gene SARS-CoV-2.
6. Cosa possiamo imparare dal tasso di mutazione del virus?
Studiando l’evoluzione virale, i ricercatori usano comunemente due misure che descrivono il tasso di cambiamento genomico. Il primo è il tasso evolutivo, che è definito come il numero medio di sostituzioni che si fissano ogni anno nei ceppi del virus, espresso in unità di mutazioni per sito all’anno. Il secondo è il tasso di mutazione, che è il numero di sostituzioni per sito per ciclo di replica. Come possiamo mettere in relazione questi due valori? Prendi in considerazione un singolo sito alla fine di un anno. L’unica misurazione di un tasso di mutazione in un β-coronavirus suggerisce che questo sito accumulerà ~ 10–6 mutazioni in ciascun ciclo di replicazione. Ogni ciclo di replica richiede ~ 10 ore e quindi ci sono 103 cicli / anno. Moltiplicando il tasso di mutazione per il numero di repliche, assumendo la neutralità e trascurando gli effetti della selezione evolutiva, arriviamo a 10–3 mutazioni per sito all’anno, coerentemente con il tasso evolutivo inferito dai genomi del coronavirus sequenziato. Poiché la nostra stima è coerente con il tasso misurato, deduciamo che il virus subisce una replica quasi continua in natura, generando costantemente nuove mutazioni che si accumulano nel corso dell’anno. Usando la nostra conoscenza del tasso di mutazione, possiamo anche trarre conclusioni su singole infezioni. Ad esempio, poiché il tasso di mutazione è ~ 10–6 mutazioni / sito / ciclo e un ml di espettorato potrebbe contenere fino a 107 RNA virali, deduciamo che ogni sito è mutato più di una volta in tali campioni.
Quanto è stabile e contagioso il virione sulle superfici?
7- Per capire come può essere trasmesso SARS-CoV-2, è di vitale importanza caratterizzare la stabilità dei virioni infettivi su diversi tipi di superfici come cartone, plastica e vari metalli. Questa è un’area molto attiva della ricerca attuale. Tuttavia, ci sono avvertenze significative associate alle misurazioni della stabilità virale. La stabilità misurata dipende dalla quantità misurata, ad esempio, si possono misurare virioni infettivi o copie virali di RNA. Il numero di virioni infettivi è in genere molto più basso di quanto dedotto dalle misurazioni del genoma virale (Woelfel et al., 2020). L’RNA SARS-CoV-2 è stato rilevato su varie superfici diverse settimane dopo il loro ultimo contatto (Moriarty et al., 2020), ma l’infezione sembra degradare più rapidamente dell’RNA. Quando i ricercatori hanno misurato la stabilità dei virioni infettivi sulle superfici, i numeri dipendevano fortemente dal tipo di superficie e dal mezzo che trasportava il virus, con la stabilità sulla plastica molto maggiore rispetto al rame o all’acciaio, ad esempio. È noto anche che la stabilità virale dipende fortemente dalla temperatura e dall’umidità (Chin et al., 2020). Pertanto, calcolare la probabilità di infezione umana dall’esposizione a superfici contaminate è un compito complesso per il quale non sono ancora disponibili dati sufficienti. Pertanto, è necessario adottare precauzioni e misure di protezione. Per ottenere qualche intuizione sull’importanza della trasmissione superficiale, consideriamo una persona infettiva non diagnosticata che tocca le superfici decine di volte durante il periodo infettivo. Prima del blocco, queste superfici pubbliche verranno successivamente toccate da centinaia di altre persone. Dal numero di riproduzione di base R0 ≈ 2–4 possiamo dedurre che non tutte le persone che toccano quelle superfici saranno infette. Limiti più dettagliati sul rischio di infezione da toccare le superfici attendono con urgenza lo studio.
Fonte: eLifeSciences

