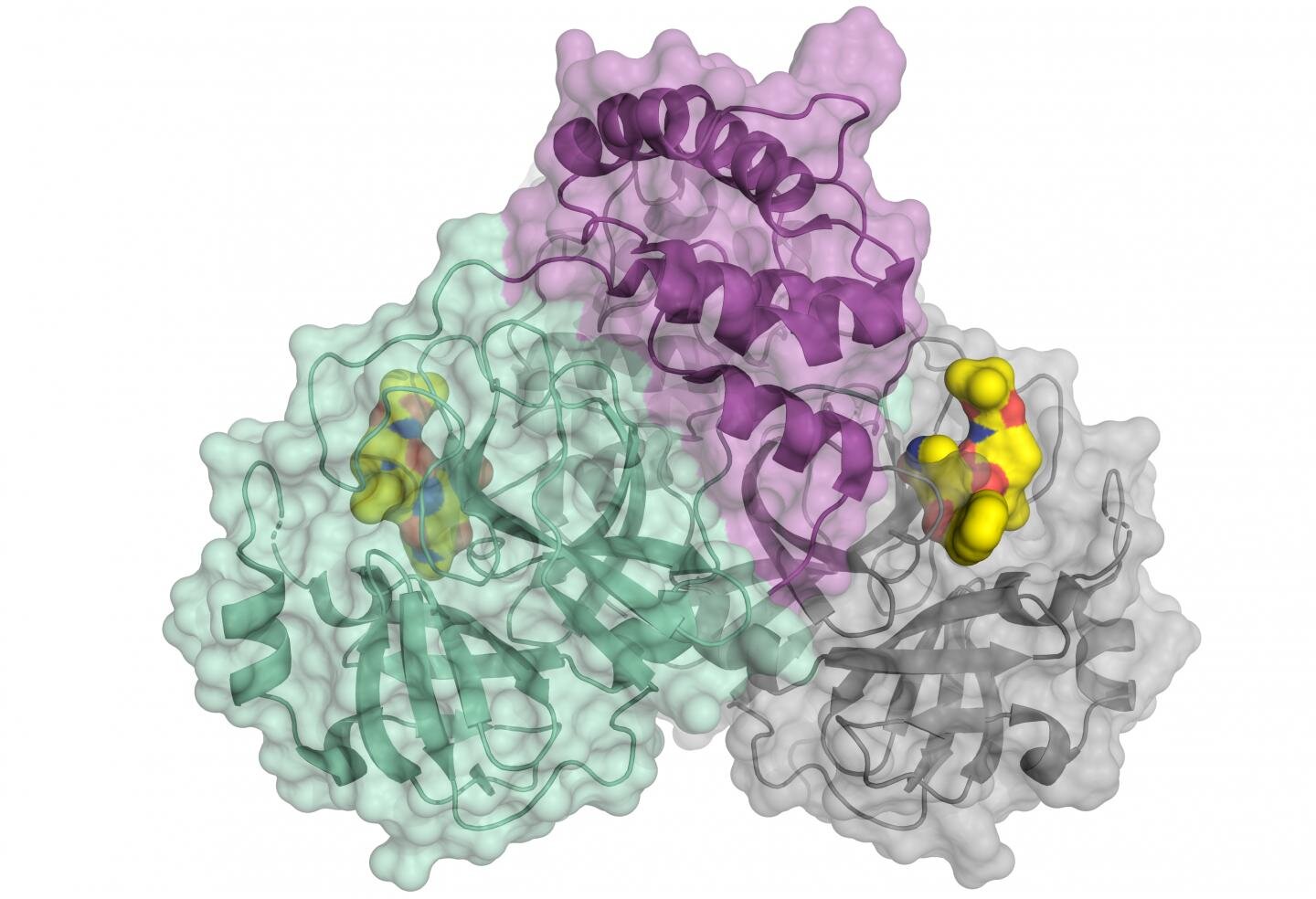
Immagine: rappresentazione schematica della proteasi coronavirus. L’enzima si presenta come un dimero costituito da due molecole identiche. Una parte del dimero è mostrata a colori (verde e viola), l’altra in grigio. La piccola molecola in giallo si lega al centro attivo della proteasi e potrebbe essere utilizzata come modello per un inibitore. © HZB
Un coronavirus sta mantenendo il mondo in sospeso. E’ SARS-CoV-2 ; è altamente infettivo e può causare una polmonite grave con difficoltà respiratoria (COVID-19). Gli scienziati stanno facendo ricerche per prevenire la replicazione del virus.
Un team dell’Università di Lubecca ha ora trovato un approccio promettente. Utilizzando la luce a raggi X ad alta intensità proveniente dalla sorgente di sincrotrone di Berlino BESSY II, i ricercatori hanno decodificato l’architettura tridimensionale della proteasi principale di SARS-CoV-2. Questa proteina è coinvolta nella riproduzione del virus. L’analisi della sua architettura 3D consente lo sviluppo sistematico di farmaci che inibiscono la riproduzione del virus.
Una proteina speciale è responsabile della riproduzione dei virus: la principale proteasi virale (Mpro o anche 3CLpro). Una team guidato dal Prof. Dr. Rolf Hilgenfeld, Università di Lubecca, ha ora decodificato l’architettura 3D della proteasi principale di SARS-CoV-2. I ricercatori hanno utilizzato la luce a raggi X ad alta intensità della struttura BESSY II dell’Helmholtz-Zentrum di Berlino.
Vedi anche: Scoperto nuovo target farmacologico per SARS-CoV-2
“Per tali questioni di massima rilevanza, possiamo offrire un accesso rapido ai nostri strumenti”, afferma il Dott. Manfred Weiss, a capo del gruppo di ricerca sulla cristallografia macromolecolare (MX) presso HZB. Ai cosiddetti strumenti MX è possibile analizzare minuscoli cristalli proteici con una luce a raggi X estremamente brillante. Le immagini contengono informazioni sull’architettura 3-D delle molecole proteiche. La forma complessa della molecola proteica e la sua densità elettronica viene quindi calcolata mediante algoritmi informatici.
“L’ architettura 3-D fornisce punti di partenza concreti per lo sviluppo di sostanze attive o inibitori. Questi farmaci potrebbero attraccare specificamente ai punti bersaglio della macromolecola e ostacolarne la funzione”, ha spiegato Rolf Hilgenfeld, un esperto di fama mondiale nel campo della virologia che ha già sviluppato un inibitore contro il virus SARS durante la pandemia di SARS del 2002/2003. Nel 2016, è riuscito a decifrare un enzima del virus Zika.
Fonte: HZB