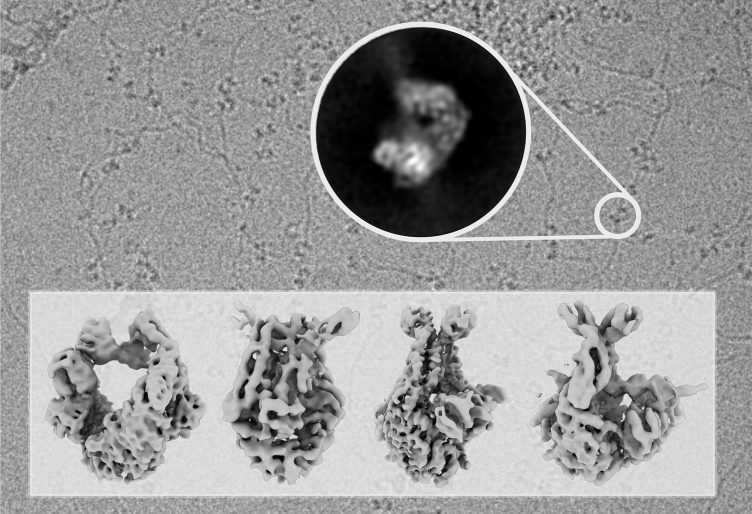
(Guardiano del genoma-Immagine: lo sfondo dell’illustrazione è una fotografia scattata con il microscopio elettronico che mostra molecole di DNA decorate con molecole MutS, che scansionano il DNA per errori. La parte inferiore mostra le strutture MutS nelle diverse fasi del processo di riparazione risolto in questo studio. / CNIO).
Gli scienziati del Genomic Integrity and Structural Biology Group, guidati da Rafael Fernández-Leiro presso il Centro nazionale spagnolo di ricerca sul cancro (CNIO), hanno scoperto come alcune proteine assicurano la riparazione degli errori introdotti nel DNA durante la sua replicazione. Usando la microscopia crioelettronica, hanno visualizzato la proteina MutS, nota anche come “guardiana del nostro genoma”. Ciò ha permesso loro di descrivere come questa singola proteina è in grado di coordinare il processo essenziale di riparazione del DNA dall’inizio alla fine.
Lo studio è stato condotto in collaborazione con Meindert Lamers del Leiden University Medical Center (LUMC, Paesi Bassi) e Titia Sixma del Netherlands Cancer Institute e dell’Oncode Institute. I loro risultati sono stati pubblicati su Nature Structural & Molecular Biology.
Una delle fasi della divisione cellulare è la replicazione del DNA, durante la quale la DNA polimerasi duplica le informazioni genetiche della cellula in modo che possa essere trasferita alla cellula figlia. Sebbene questo sia un processo molto preciso, a volte si verificano degli errori. È essenziale che questi errori vengano riparati, poiché altrimenti potrebbero portare allo sviluppo di tumori.
Vedi anche:Nel genoma gli indizi sulla longevità umana
I ricercatori avevano già descritto in precedenti pubblicazioni che la DNA polimerasi ha un proprio riparatore, un’esonucleasi che le consente di correggere gli errori che vengono introdotti durante la replicazione del DNA. Ma quando questo correttore non è sufficiente, entra in gioco la proteina MutS che scansiona il DNA copiato alla ricerca di errori, quindi avvia e completa la riparazione di eventuali errori rilevati. Ma fino ad ora, non era chiaro come una singola proteina potesse coordinare così tanti processi diversi. Lo studio internazionale ora pubblicato è riuscito a svelare il meccanismo.
“Utilizzando la microscopia crioelettronica, siamo stati in grado di osservare MutS mentre svolgeva le sue funzioni e catturare la sua struttura molecolare in conformazioni successive. Con queste informazioni, siamo stati in grado di capire come una singola proteina può coordinare l’intero processo che deve essere estremamente accurato”, spiega Rafael Fernández-Leiro.
Una conoscenza approfondita del processo di riparazione del DNA, in cui sono coinvolte la DNA polimerasi, l’esonucleasi e la proteina MutS, è essenziale per capire come le alterazioni in una qualsiasi di queste proteine portino a mutazioni e, quindi, ad un aumentato rischio di sviluppare alcuni tipi di tumore, come la sindrome di Lynch e il cancro dell’endometrio.
I ricercatori sottolineano che il disfacimento delle strutture proteiche è possibile solo grazie agli enormi progressi tecnologici nella microscopia elettronica negli ultimi anni. “La microscopia elettronica ci permette di ottenere immagini ad altissima risoluzione delle proteine mentre svolgono le loro funzioni. Con queste immagini, possiamo ricostruire la struttura tridimensionale della proteina al computer e generare un modello atomico che ci permette di capire come funziona “, continua Fernández-Leiro.
Spiegano gli autori:
“La riparazione del disadattamento del DNA rileva e rimuove i disallineamenti dal DNA mediante un meccanismo conservato, riducendo il tasso di errore di replicazione del DNA da 100 a 1.000 volte. In questo processo, gli omologhi MutS scansionano il DNA, riconoscono le mancate corrispondenze e avviano la riparazione. Non è chiaro come gli omologhi MutS autorizzino selettivamente la riparazione di una mancata corrispondenza tra milioni di coppie di basi abbinate. Qui presentiamo quattro strutture crio-EM di Escherichia coliMutS che forniscono istantanee, dalla scansione del DNA omoduplex al legame non corrispondente e all’attivazione di MutL attraverso uno stato intermedio. Durante la scansione, il DNA omoduplex forma un blocco sterico che impedisce a MutS di passare allo stato di morsetto legato a MutL, che può essere superato solo attorcigliando il DNA in caso di mancata corrispondenza. L’asimmetria strutturale in tutte e quattro le strutture indica una divisione del lavoro tra i due monomeri MutS. Insieme, queste strutture rivelano come un piccolo cambiamento conformazionale da MutS legato da omoduplex a eteroduplex agisce come un passaggio di licenza che innesca un drammatico cambiamento conformazionale che consente il legame MutL e l’inizio della cascata di riparazione“.
Fonte:Nature