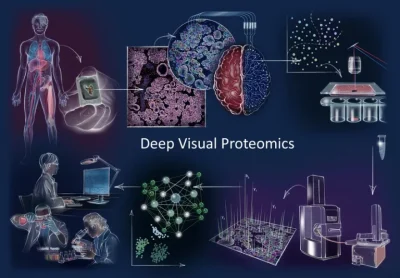
Cancro-immagine: concetto e flusso di lavoro di Deep Visual Proteomics In senso orario: Deep Visual Proteomics (DVP) combina imaging ad alta risoluzione, analisi delle immagini guidata dall’intelligenza artificiale (AI) per la classificazione e l’isolamento di singole cellule con un nuovo flusso di lavoro di proteomica ultra sensibile. DVP collega l’imaging ricco di dati di colture cellulari o tessuti archiviati di biobanche di pazienti con segmentazione cellulare basata sull’apprendimento profondo e identificazione basata sull’apprendimento automatico di tipi e stati cellulari. Oggetti di interesse cellulari o subcellulari classificati dall’AI (non) supervisionati vengono sottoposti a microdissezione laser automatizzata e profilazione proteomica basata sulla spettrometria di massa (MS). La successiva analisi dei dati bioinformatici consente il data mining per scoprire firme proteiche che forniscono approfondimenti molecolari sulla variazione del proteoma negli stati di salute e malattia a livello di singole cellule. Credito immagine: MPI of Biochemistry
Come nasce il cancro? In che modo la composizione cellulare influenza la malignità del tumore? Queste domande sono profonde e difficili, ma sono cruciali per comprendere la malattia e trovare la cura giusta.
Ora, un team tedesco-danese guidato da Matthias Mann ha sviluppato una tecnologia rivoluzionaria chiamata “Deep Visual Proteomics“. Questo metodo fornisce ai ricercatori e ai clinici una lettura proteica per comprendere il cancro con una risoluzione di tipo cellula singola. La tecnologia dimostra il suo potenziale nella prima applicazione alle cellule tumorali.
“Quando qualcosa va storto all’interno delle nostre cellule e ci ammaliamo, puoi star certo che le proteine sono coinvolte in un’ampia gamma di modi diversi. Per questo motivo, la mappatura del panorama proteico può aiutarci a determinare perché un tumore potrebbe svilupparsi in un particolare paziente, quali vulnerabilità ha quel tumore e anche quale strategia di trattamento potrebbe rivelarsi la più vantaggiosa”, spiega Matthias Mann del Max Planck Institute of Biochemistry vicino a Monaco e del Novo Nordisk Foundation Center for Protein research presso l’Università di Copenaghen.
Incuriosito da queste domande, un team di scienziati multidisciplinari dei gruppi di ricerca guidati da Matthias Mann ha inventato un metodo innovativo chiamato “Deep Visual Proteomics”. Lo studio combina le caratteristiche visive di un tumore con una tecnologia di profilazione profonda per visualizzare le firme proteiche nelle cellule aberranti adiacenti alle cellule sane circostanti. Ciò fornirebbe ai ricercatori una visione senza precedenti della malattia e guiderebbe gli oncologi nello sviluppo di strategie intelligenti per la diagnosi e il trattamento.
Deep Visual Proteomics unisce quattro tecnologie
Deep Visual Proteomics integra per la prima volta i progressi di quattro diverse tecnologie in un unico flusso di lavoro: in primo luogo, la microscopia avanzata genera mappe tissutali ad alta risoluzione. In secondo luogo, algoritmi di apprendimento automatico e intelligenza artificiale vengono utilizzati per classificare accuratamente le cellule in base a forma, dimensione o localizzazione proteica prima che le singole cellule vengano raccolte tramite microdissezione laser ad alta precisione.
Quindi, dopo aver selezionato cellule normali o di diverso tipo malate, le migliaia di proteine presenti nelle popolazioni cellulari vengono rilevate tutte in una volta utilizzando strumenti di spettrometria di massa ad altissima sensibilità da questa minuscola quantità di campione. Infine, sofisticate analisi bioinformatiche generano mappe di proteine che forniscono una risoluzione spaziale delle firme proteiche in malattie altamente complesse come il cancro. Tali paesaggi proteici sono strumenti preziosi per i medici nella comprensione dei meccanismi nella salute e nella malattia in modo più dettagliato.
“Il nostro nuovo concetto, ‘Deep Visual Proteomics’, potrebbe diventare un punto di svolta per la patologia molecolare negli ospedali. Con questo metodo, prendiamo un campione di tessuto con cellule tumorali e possiamo identificare e determinare migliaia di proteine in un minuto di tempo e sforzo. Queste firme proteomiche rivelano i meccanismi che guidano lo sviluppo del tumore ed espongono direttamente nuovi bersagli terapeutici da una singola fetta di tessuto di una biopsia di un paziente oncologico. Espone un cosmo di molecole all’interno di queste cellule tumorali”, afferma Andreas Mund, Professore associato presso il Center for Protein Research e parte del team del Professor Matthias Mann, che ha guidato questo sviluppo.
Rilevanza per la patologia clinica
Nel nuovo studio pubblicato da Nature Biotechnology, i ricercatori hanno applicato la ‘Deep Visual Proteomics’ alle cellule di pazienti con carcinoma a cellule aciniche e con melanoma. Lise Mette Rahbek Gjerdrum, consulente e Professore associato di ricerca clinica presso il Dipartimento di Patologia, Zealand University Hospital e Dipartimento di Medicina Clinica, Università di Copenaghen, descrive: “Questo metodo unico combina l’architettura dei tessuti con l’espressione di migliaia di proteine specifiche per cellule selezionate. Consente ai ricercatori di studiare le interazioni tra le cellule tumorali e le cellule circostanti con importanti implicazioni per il futuro trattamento clinico del cancro. Di recente, abbiamo diagnosticato un caso clinico altamente complesso utilizzando la ‘Deep Visual Proteomics’ “.
Fabian Coscia, coautore principale dello studio e, da giugno 2021, responsabile del gruppo di ricerca “Spatial Proteomics” presso il Max Delbrück Center for Molecular Medicine dell’Associazione Helmholtz di Berlino, afferma: “Ma naturalmente la tecnica può essere utilizzata per caratterizzare tutti gli altri tipi di tumore in dettagli simili”. Il suo obiettivo”, afferma, “è quello di attingere alle biobanche dei pazienti archiviate e trovare nuovi obiettivi per terapie antitumorali personalizzate, ovvero personalizzate per il paziente, anche per tumori precedentemente resistenti al trattamento”.
E non è solo il cancro a poter essere meglio compreso usando la ‘Deep Visual Proteomics’. Il metodo può essere applicato anche ad altre malattie.
Leggi anche:Farmaci del futuro potrebbero interrompere la filiera che alimenta il cancro al seno
“Ad esempio, puoi analizzare le proteine nelle cellule nervose per scoprire esattamente cosa succede nelle cellule nelle malattie neurodegenerative come l’Alzheimer o il Parkinson“, afferma ulteriormente Fabian Coscia. ”Unendo la potenza della microscopia, dell’intelligenza artificiale e della proteomica basata sulla spettrometria di massa ultra sensibile, abbiamo sviluppato un metodo molto potente per comprendere il cablaggio molecolare delle cellule sane rispetto a quelle malate. Ciò potrebbe aiutare i medici a identificare i target per farmaci e diagnosi future’‘, conclude Matthias Mann.
Fonte: MPG