SARS-CoV-2 -immagine credit public domain.
Dopo 150.000 articoli e 17 milioni di sequenze del genoma, cosa ci ha insegnato la scienza sul SARS-CoV-2?
Kei Sato era alla ricerca della sua prossima grande sfida cinque anni fa, quando questa si è abbattuta su di lui e sul mondo intero. Il virologo aveva da poco fondato un gruppo indipendente presso l’Università di Tokyo e stava cercando di ritagliarsi una nicchia nell’affollato campo della ricerca sull’HIV. “Ho pensato: ‘Cosa posso fare per i prossimi 20 o 30 anni?‘ “, dice.
Ha trovato una risposta nel SARS-CoV-2, il virus responsabile della pandemia di COVID-19, che si stava rapidamente diffondendo in tutto il mondo. A marzo 2020, mentre circolavano voci secondo cui Tokyo avrebbe potuto affrontare un lockdown che avrebbe fermato le attività di ricerca, Sato e cinque studenti si sono trasferiti nel laboratorio di un ex consulente a Kyoto. Lì, hanno iniziato a studiare una proteina virale che il SARS-CoV-2 usa per reprimere le prime risposte immunitarie del corpo. Sato ha presto fondato un consorzio di ricercatori che avrebbe pubblicato almeno 50 studi sul virus.
In soli cinque anni, il SARS-CoV-2 è diventato uno dei virus più esaminati del pianeta. I ricercatori hanno pubblicato circa 150.000 articoli di ricerca al riguardo, secondo il database di citazioni Scopus. Si tratta di circa tre volte il numero di articoli pubblicati sull’HIV nello stesso periodo. Gli scienziati hanno anche generato finora più di 17 milioni di sequenze del genoma del SARS-CoV-2, più che per qualsiasi altro organismo. Ciò ha fornito una visione senza pari sui modi in cui il virus è cambiato con la diffusione delle infezioni. “C’era l’opportunità di vedere una pandemia in tempo reale con una risoluzione molto più elevata di quanto fosse mai stato possibile prima“, afferma Tom Peacock, virologo presso il Pirbright Institute, vicino a Woking, Regno Unito.
Ora, con la fase di emergenza della pandemia ormai alle spalle, i virologi stanno facendo il punto su ciò che si può imparare su un virus in così poco tempo, inclusa la sua evoluzione e le sue interazioni con gli ospiti umani. Ecco quattro lezioni tratte dalla pandemia che alcuni affermano potrebbero potenziare la risposta globale alle future pandemie, ma solo se istituzioni scientifiche e di sanità pubblica saranno pronte a usarle.
Le sequenze virali raccontano storie
L’11 gennaio 2020, Edward Holmes, virologo dell’Università di Sydney, in Australia, ha condiviso quella che la maggior parte degli scienziati considera la prima sequenza del genoma SARS-CoV-2 su un forum di discussione di virologia; aveva ricevuto i dati dal virologo Zhang Yongzhen in Cina.
Entro la fine dell’anno, gli scienziati avevano inviato più di 300.000 sequenze a un archivio noto come Global Initiative on Sharing All Influenza Data (GISAID). Da lì in poi, il tasso di raccolta dati è solo aumentato man mano che le varianti preoccupanti del virus prendevano piede. Alcuni paesi hanno investito enormi risorse nel sequenziamento del SARS-CoV-2: tra loro, il Regno Unito e gli Stati Uniti hanno contribuito con più di 8,5 milioni (vedi “Viral genome rally”). Nel frattempo, gli scienziati di altri paesi, tra cui Sudafrica, India e Brasile, hanno dimostrato che una sorveglianza efficiente può individuare varianti preoccupanti in contesti con risorse inferiori.
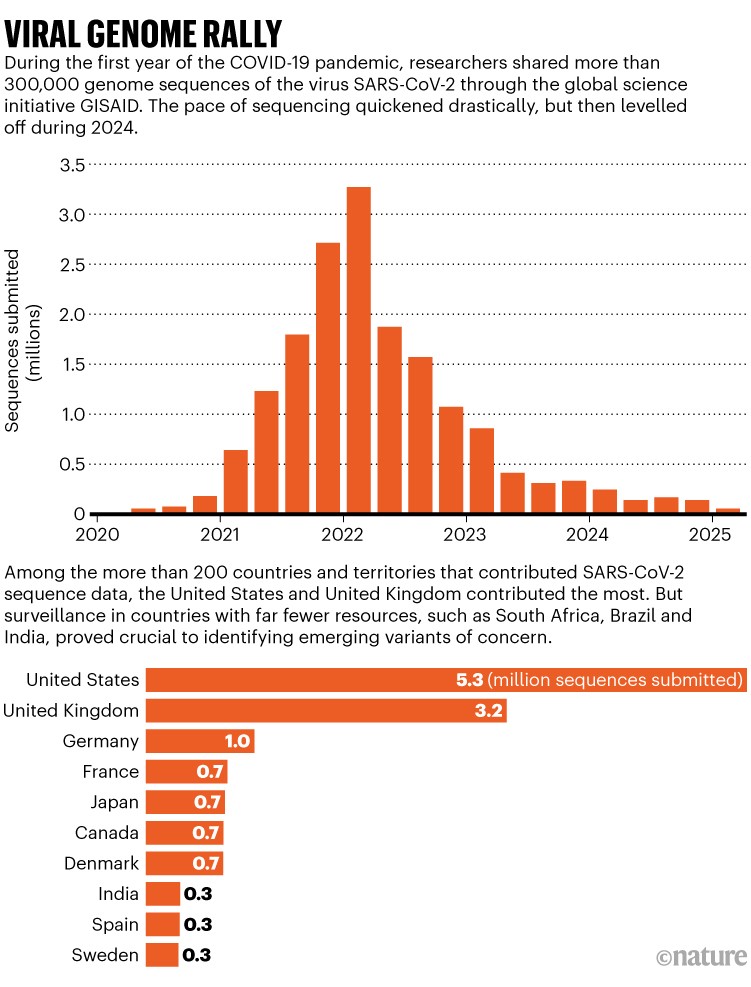
Fonte: GISAID
Nelle epidemie precedenti, come quella di Ebola nell’Africa occidentale del 2013-16, i dati di sequenziamento arrivavano troppo lentamente per tracciare come il virus stava cambiando con la diffusione delle infezioni. “Ma è diventato subito chiaro che le sequenze di SARS-CoV-2 sarebbero arrivate a un volume e a un ritmo senza precedenti”, afferma Emma Hodcroft, epidemiologa genomica presso lo Swiss Tropical and Public Health Institute di Basilea. Hodcroft lavora a un’iniziativa chiamata Nextstrain, che utilizza i dati del genoma per tracciare i virus, come l’influenza, per comprenderne meglio la diffusione. “Avevamo sviluppato così tanti di questi metodi che, in teoria, avrebbero potuto essere molto utili“, afferma Hodcroft. “E all’improvviso, nel 2020, abbiamo avuto l’opportunità di presentarci e farci vedere“.
Inizialmente, i dati di sequenziamento del SARS-CoV-2 sono stati utilizzati per tracciare la diffusione del virus nel suo epicentro a Wuhan, in Cina e poi a livello globale. Ciò ha risposto alle prime domande chiave, come se il virus si fosse diffuso in gran parte tra le persone o dalle stesse fonti animali agli esseri umani. I dati hanno rivelato le rotte geografiche attraverso cui viaggiava il virus e le hanno mostrate molto più rapidamente di quanto potessero fare le indagini epidemiologiche convenzionali. Successivamente, hanno iniziato ad apparire varianti del virus a trasmissione più rapida, che hanno mandato i laboratori di sequenziamento in ipervelocità. Un collettivo globale di scienziati e dilettanti che tracciano le varianti ha scandagliato costantemente i dati di sequenza alla ricerca di preoccupanti cambiamenti virali.
“È diventato possibile tracciare l’evoluzione di questo virus in modo estremamente dettagliato per vedere esattamente cosa stava cambiando”, afferma Jesse Bloom, un biologo evoluzionista virale presso il Fred Hutchinson Cancer Center di Seattle, Washington. Con milioni di genomi SARS-CoV-2 in mano, i ricercatori possono ora tornare indietro e studiarli per comprendere i vincoli all’evoluzione del virus. “È qualcosa che non siamo mai stati in grado di fare prima“, afferma Hodcroft.
I virus cambiano più del previsto
Poiché nessuno aveva mai studiato prima il SARS-CoV-2, gli scienziati hanno avanzato le proprie ipotesi su come si sarebbe adattato. Molti sono stati guidati dalle esperienze con un altro virus RNA che causa infezioni respiratorie: l’influenza. “Non avevamo molte informazioni su altri virus respiratori che avrebbero potuto causare pandemie”, afferma Hodcroft.
L’influenza si diffonde principalmente tramite l’acquisizione di mutazioni che le consentono di eludere l’immunità delle persone. Poiché nessuno era mai stato infettato dal SARS-CoV-2 prima del 2019, molti scienziati non si aspettavano di vedere grandi cambiamenti virali fino a quando non ci fosse stata una pressione sostanziale esercitata su di esso dal sistema immunitario delle persone, tramite infezioni o, meglio ancora, vaccinazione.
L’emergere di varianti più veloci e letali del SARS-CoV-2, come Alpha e Delta, ha cancellato alcune delle prime ipotesi. Anche all’inizio del 2020, il SARS-CoV-2 aveva acquisito un singolo cambiamento di amminoacido che ne aveva notevolmente aumentato la diffusione. Molti altri ne avrebbero seguito l’esempio.
“Quello che ho sbagliato e che non avevo previsto era quanto sarebbe cambiato fenotipicamente il virus”, dice Holmes. “Si è vista questa incredibile accelerazione nella trasmissibilità e nella virulenza. Ciò suggerisce che il SARS-CoV-2 non era particolarmente adatto a diffondersi tra le persone quando è emerso a Wuhan, una città di milioni di persone. Avrebbe potuto benissimo esaurirsi in un contesto meno densamente popolato”.
Holmes si chiede anche se il ritmo vertiginoso del cambiamento osservato sia stato semplicemente il prodotto di quanto attentamente è stato monitorato il SARS-CoV-2. I ricercatori vedrebbero lo stesso tasso se osservassero l’emergere di un ceppo influenzale nuovo per la popolazione, alla stessa risoluzione? Questo resta da stabilire.
I primi passi da gigante compiuti da SARS-CoV-2 sono stati accompagnati da una nota positiva: non hanno influenzato drasticamente l’immunità protettiva fornita dai vaccini e dalle infezioni precedenti. Ma ciò è cambiato con l’emergere della variante Omicron alla fine del 2021, che era carica di cambiamenti alla sua proteina “spike” che l’hanno aiutata a schivare le risposte anticorpali (la proteina spike consente al virus di entrare nelle cellule ospiti). Scienziati come Bloom sono rimasti sbalorditi dalla rapidità con cui questi cambiamenti sono apparsi nelle successive varianti post-Omicron.
“E questo non è stato nemmeno l’aspetto più sorprendente di Omicron”, afferma Ravindra Gupta, virologo presso l’Università di Cambridge, Regno Unito. Poco dopo l’emergere della variante, il suo team e altri hanno notato che, a differenza delle precedenti varianti di SARS-CoV-2 come Delta che favorivano le cellule delle vie aeree inferiori del polmone, Omicron preferiva infettare le vie aeree superiori. “Documentare che un virus ha cambiato il suo comportamento biologico nel corso di una pandemia è stato senza precedenti“, afferma Gupta.
La preferenza di Omicron per le vie aeree superiori ha probabilmente contribuito alla sua mitezza clinica, ovvero alla sua virulenza relativamente bassa, rispetto alle iterazioni precedenti. “Ma è difficile districare questo cambiamento dal fatto che Omicron ha colpito dopo che gran parte del mondo aveva iniziato a stabilire una certa immunità”, afferma Bloom, “e ci sono prove che Omicron fosse cattivo quanto la versione di SARS-CoV-2 emersa a Wuhan“.
E sebbene Omicron e i suoi derivati fossero più blandi di Alpha, Beta e Delta, questi si erano dimostrati tutti più virulenti della linea che avevano sostituito, rovesciando l’idea che il virus si sarebbe evoluto per essere meno letale. “L’idea che ci sia una qualche legge della natura che dice che un virus perderà rapidamente la sua virulenza quando salta in un nuovo ospite è errata“, afferma Bloom. “È un’idea che non ha mai avuto molto consenso tra i virologi, comunque“.
Uno dei grandi timori di Sato è che emerga una variante drasticamente diversa del SARS-CoV-2 che superi l’immunità che impedisce alla maggior parte delle persone di ammalarsi gravemente. Teme che il risultato potrebbe essere disastroso.
I casi cronici potrebbero rivelare intuizioni
Prima che Gupta rivolgesse la sua attenzione al SARS-CoV-2, il suo focus era sull’HIV, che di solito è un’infezione che dura tutta la vita. Come medico, aveva curato la seconda persona mai guarita dall’HIV tramite un trapianto di cellule staminali del sangue. Ma il suo gruppo di ricerca ha studiato come la resistenza ai farmaci antiretrovirali si evolve nel corso di mesi e anni nelle persone.
La maggior parte degli scienziati ha presunto che, a differenza dell’HIV o di altre infezioni a lungo termine, i virus respiratori come il SARS-CoV-2 fossero acuti e che coloro che sopravvivevano alle infezioni eliminavano il virus nel giro di pochi giorni. Le infezioni a lungo termine si verificano nell’influenza, ma sembrano essere un vicolo cieco evolutivo. Il virus si adatta per sopravvivere nell’ospite, non per diffondersi ad altri.

Gli scienziati di Nuova Delhi preparano campioni di coronavirus per il sequenziamento. Credito: T. Narayan/Bloomberg tramite Getty Images.
Ma alla fine del 2020, Gupta ha caratterizzato un’infezione da SARS-CoV-2 di 102 giorni in un uomo di 70 anni con un sistema immunitario compromesso. L’infezione è stata alla fine fatale. Nel corpo dell’uomo, il virus ha sviluppato un numero elevato di cambiamenti nella proteina spike. Molti di questi sarebbero stati osservati anche in varianti preoccupanti, tra cui la variante Alpha che ha fatto schizzare alle stelle i conteggi dei casi e ha provocato un’altra ondata di lockdown alla fine del 2020 e all’inizio del 2021.
Leggi anche:SARS-CoV-2: le proteine spike persistono nel cervello per anni
Il caso dell’uomo non ha dato origine a nessuna variante diffusa, ma ha dato a Gupta, con il suo background nell’evoluzione dell’HIV, l’idea che le infezioni croniche potessero essere una fonte dei drastici balzi evolutivi che hanno caratterizzato le varianti preoccupanti del SARS-CoV-2. “Non avevamo i preconcetti che il campo dell’influenza aveva su cosa fanno i virus respiratori”, dice.
Alex Sigal, un virologo dell’Africa Health Research Institute di Durban, in Sudafrica, ha avuto un’idea simile quando un’altra variante, chiamata Beta, è stata identificata nel suo Paese. Il Sudafrica ha un alto tasso di infezioni da HIV, molte delle quali non vengono curate e Sigal si è chiesto se fosse più di una coincidenza che Beta sembrasse essere emersa dove c’erano un numero elevato di persone immunodepresse.
Fonte:Nature