(RNA-Immagine: astratto grafico. Credito: Terapia Molecolare – Acidi Nucleici (2022). DOI: 10.1016/j.omtn.2022.08.031).
L’RNA è spesso descritto come il cugino a filamento singolo del DNA, la molecola a doppio filamento che costituisce il genoma di tutti gli organismi viventi. Molti tipi di virus tuttavia, trasportano le loro informazioni genetiche in una forma di RNA a doppio filamento (dsRNA). Quando il sistema immunitario umano rileva la presenza di questo RNA estraneo, suona l’allarme aumentando la produzione di citochine protettive dette interferoni (IFN), che attivano la risposta immunitaria innata contro gli invasori virali.
Gli scienziati hanno tentato di sfruttare questa risposta antivirale naturale creando dsRNA terapeutici che imitano le caratteristiche dei genomi virali. Ma l’ingerenza nel sistema immunitario innato è un’arma a doppio taglio. Gli analoghi dell’RNA virale possono anche attivare percorsi molecolari che portano a un’eccessiva infiammazione nel corpo, che potrebbero causare più danni dei loro benefici antivirali.
Ora, un gruppo di ricercatori del Wyss Institute for Biologically Inspired Engineering dell’Università di Harvard ha scoperto una nuova classe di dsRNA immunostimolatori che induce potentemente la produzione di due forme di IFN (IFN-I e IFN-III) limitando l’infiammazione comunemente osservata con i precedenti tipi di immunostimolanti a base di RNA.
Le nuove molecole di dsRNA hanno inibito drasticamente la capacità di molti virus con potenziale pandemico, inclusi SARS-CoV, SARS-CoV-2, MERS-CoV e ceppi multipli di influenza A, di infettare le cellule umane, sia nella coltura cellulare tradizionale che in quella modelli complessi di chip d’organo umano del polmone. In un modello murino di COVID-19, il dsRNA ha ridotto la quantità di virus nei corpi degli animali di oltre 1.000 volte.
I risultati dello studio sono stati pubblicati oggi in Molecular Therapy—Nucleic Acids.
“Questi nuovi dsRNA sono un’opzione di trattamento interessante per COVID-19 perché l’infezione da SARS-CoV-2 produce una risposta immunitaria squilibrata in cui la reazione protettiva dell’IFN viene soppressa mentre la reazione infiammatoria è elevata. Aumentando preferenzialmente l’IFN-I, il nostro dsRNA ha il potenziale per correggere questo squilibrio e potrebbe essere utilizzato anche per trattare molte altre malattie virali”, ha affermato il co-primo autore Haiqing Bai, Ph.D., ex Wyss Technology Development Fellow che ora è Direttore dello sviluppo preclinico presso Xellar Biosystems.
Scoperta fortuita, investigazione sistematica
Per anni, Bai e altri membri del laboratorio del Direttore fondatore di Wyss, Don Ingber, hanno sviluppato nuovi trattamenti per le malattie virali utilizzando la loro piattaforma umana Organ Chip. Prima del 2020, il team stava lavorando all’identificazione dei geni nelle cellule polmonari umane che regolano le loro risposte all’infezione da influenza A, che all’epoca era il virus di maggiore preoccupazione per la pandemia.
I ricercatori hanno iniziato lo studio utilizzando lo screening CRISPR-Cas9 per identificare i geni ospiti che potrebbero inibire la proliferazione del virus dell’influenza. Uno dei loro migliori candidati allo screening era un gene chiamato DGCR5, che produce una lunga sequenza di RNA non codificante piuttosto che una proteina. Per vedere in che modo DGCR5 ha influenzato i tassi di infezione, i ricercatori hanno “abbattuto” il gene progettando tre diversi piccoli RNA interferenti a doppio filamento (siRNA) per interferire con l’espressione del gene.
I ricercatori hanno scoperto che questa miscela ha ridotto i livelli di DGCR5 di oltre l’80% e ha inibito l’infezione da influenza A di oltre il 90%. Eccitati da questi risultati, hanno iniziato a testare i siRNA individualmente e alcune sequenze di siRNA specifiche per DGCR5. Con loro sorpresa, mentre nove dei dieci siRNA che hanno creato hanno ridotto significativamente l’espressione del gene DGCR5, solo uno di loro ha inibito l’infezione virale. Sembrava che l’attività di DGCR5 potesse non essere il driver dei tassi di infezione virale.
Studiando ulteriormente, hanno confermato che questo particolare siRNA ha causato la sovraregolazione di più geni coinvolti nella via di segnalazione dell’IFN. Ulteriori studi hanno rivelato che il siRNA ha attivato esclusivamente una delle tre vie di segnalazione che possono innescare la produzione di IFN, nota come via RIG-I, che è una componente essenziale della risposta immunitaria innata. I ricercatori hanno iniziato a sospettare di aver scoperto una nuova classe di dsRNA che stimolava il sistema immunitario attraverso un meccanismo d’azione precedentemente sconosciuto.
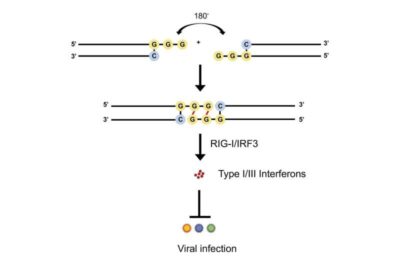
Hanno lavorato sistematicamente generando e testando più di 200 variazioni sulla sequenza originale di dsRNA per svelare quel meccanismo. Hanno identificato una particolare stringa di nucleotidi, chiamata “motivo”, che era costantemente presente su un’estremità dei dsRNA con un’elevata attività di stimolazione dell’IFN: una citosina (C) su un filamento e tre guanine (GGG) sull’altro filamento. Poiché la C si lega a una G della sequenza GGG nel motivo, c’è una “sporgenza” di due G alla fine di ciascun dsRNA. Quando sono presenti molte copie del dsRNA, le G sovrastanti di una molecola possono legarsi a quelle di un’altra molecola tramite un fenomeno insolito chiamato accoppiamento di basi GG Hoogsteen. I dimeri dsRNA risultanti si legano quindi direttamente a RIG-I in modo molto efficace, provocandone l’attivazione e la successiva risposta IFN.
“Questa scoperta è stata molto interessante per noi, perché studi precedenti avevano dimostrato che RIG-I può essere attivato da dsRNA duplex ‘smussati’ privi di sporgenze e che quasi ogni tipo di sporgenza può impedire il legame RIG-I. “Ma l’esclusivo GG per il comportamento di legame che abbiamo osservato, crea efficacemente dsRNA con estremità smussata che possono attivare potentemente RIG-I, quindi in questo caso la sporgenza è cruciale per l’attività del nostro nuovo dsRNA”, ha affermato il co-primo autore Longlong Si, Ph.D., a ex Wyss Technology Development Fellow che ora è professore allo Shenzhen Institute of Advanced Technology in Cina.
Dall’influenza al COVID e oltre
Per vedere quanto fossero efficaci i loro dsRNA scoperti di recente nelle cellule viventi, i ricercatori hanno testato uno di essi contro un immunostimolante sintetico che imita le infezioni virali, chiamato poly(I:C). Quando hanno applicato i due trattamenti alle cellule epiteliali umane, hanno scoperto che il dsRNA produceva una risposta antivirale meno infiammatoria, mentre il poli(I:C) più infiammatorio causava cambiamenti molto più ampi nell’espressione genica e influiva su altri processi biologici che sono essenziali per il normale funzione cellulare.
Facendo un ulteriore passo avanti, il team ha quindi testato il dsRNA nelle vie aeree polmonari umane e nei chip Alveolus precedentemente sviluppati presso il Wyss Institute per replicare in vitro il complesso tessuto e le funzioni degli organi umani. I ricercatori hanno introdotto il dsRNA in chip sani e hanno visto che l’espressione di IFN-I aumentava da 12 a 40 volte. Quando hanno poi aggiunto l’influenza A agli Organ Chips, hanno scoperto che il dsRNA sopprimeva l’infezione dell’80-90%.
Quando la pandemia di COVID-19 ha colpito, il team ha spostato i propri studi dall’influenza al SARS-CoV-2 e ai relativi coronavirus SARS-CoV, MERS-CoV e al virus del raffreddore comune HCoV-NL63. Il loro dsRNA ha preso il cambio di passo, inibendo l’infezione da MERS-CoV e HCoV-NL63 di una linea cellulare di mammiferi derivata da scimmie di oltre il 90% e SARS-CoV di oltre 1.000 volte. Ha anche inibito l’infezione da SARS-CoV-2 di una linea cellulare epiteliale umana di uno sbalorditivo 99,99%.
Questi studi sono stati completati in collaborazione con il gruppo di Matthew Frieman della University of Maryland School of Medicine e Benjamin tenOever della Icahn School of Medicine del Mount Sinai (ora Grossman School of Medicine della New York University).
Vedi anche:RNA: creato per la prima volta RNA che si replica
Infine, i ricercatori hanno testato il dsRNA in un modello murino di COVID-19 con i collaboratori Dong Yang e Colleen Johnsson, Ph.D. presso l’Università del Tennessee Health Science Center. Quando hanno infettato i topi trattati con SARS-CoV-2, il dsRNA ha ridotto la carica virale nei polmoni degli animali di oltre 1.000 volte rispetto agli animali trattati con una sequenza di dsRNA criptata.
“La pandemia da COVID-19 ha reso dolorosamente chiaro che abbiamo bisogno di terapie ad ampio spettro in grado di attenuare l’infezione da un’ampia varietà di virus, piuttosto che sviluppare un trattamento su misura per ogni singola malattia man mano che si manifesta. Spero che questa tecnologia terapeutica dell’RNA, le cui proprietà chimiche e fisiche la rendono facilmente realizzabile su larga scala, diventerà un approccio ampiamente utilizzato per combattere le future pandemie”, ha affermato Don Ingber, MD, Ph.D., che è anche il Judah Folkman Professor of Vascular Biologia presso la Harvard Medical School (HMS) e il Boston Children’s Hospital.
Oltre a trattare i virus, questi nuovi dsRNA potrebbero trattare infezioni batteriche, fungine e parassitarie, nonché condizioni come il cancro e malattie autoimmuni che potrebbero trarre vantaggio da una maggiore produzione di IFN. Potrebbero anche essere usati come adiuvanti per potenziare l’attività di altri vaccini.
Gli autori affermano che è importante per gli studi futuri stabilire la tempistica ottimale del trattamento, poiché l’attivazione dell’IFN troppo tardi in un’infezione può esacerbare l’infiammazione, nonché è importante studiare studiare la possibilità di somministrare il dsRNA direttamente alle vie aeree superiori (ad esempio, tramite un inalatore) per ridurre al minimo l’attivazione immunitaria sistemica.