La ribosilazione dell’ADP, un processo guidato dagli enzimi PARP, aiuta a far crescere alcuni tumori e si sospetta che sia un fattore di altre malattie comuni.
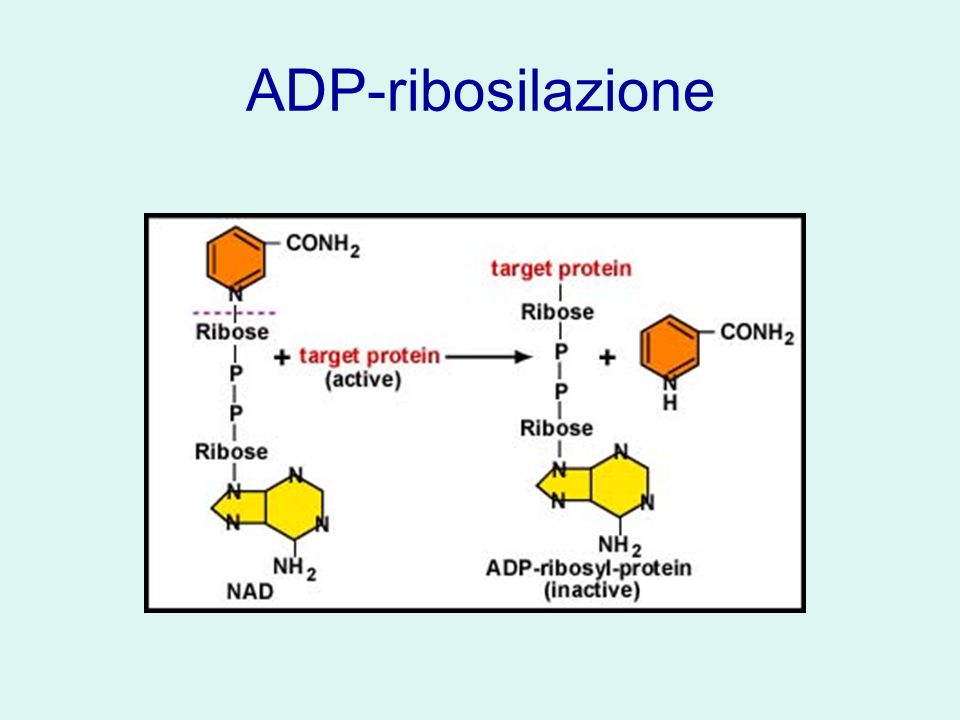
‘La ADP-ribosilazione è l’aggiunta di uno o più molecole di ADP ribosio a una proteina. L’ADP ribosio proviene da una molecole di NAD+ che viene tagliata rilasciando una molecola di Nicotinamide ad opera di enzimi eucariotici della famiglia di ADP-ribosil-transferasi (ADPRT)’.
Un nuovo metodo sviluppato dagli scienziati della Johns Hopkins Bloomberg School of Public Health probabilmente velocizzerà lo studio di questo importante processo biologico.
La ribosilazione di ADP si verifica a livelli elevati in alcuni tipi di cancro e i farmaci chiamati inibitori di PARP, che bloccano la ribosilazione di ADP, comprendono una delle nuove classi di terapia del cancro più promettenti. Eppure la ricerca in questa importante area è stata limitata dalla mancanza di metodi di laboratorio per studiare la ribosilazione dell’ADP. PARP sta per poli-ribosio polimerasi ADP.
Il nuovo metodo, descritto in un documento apparso il 31 gennaio in Molecular Cell, consente agli scienziati di fissare piccoli fari o manipoli chimici su molecole di ribosio ADP per studiare in dettaglio in che modo la ribosilazione dell’ADP colpisce le cellule in salute e malattia.
“La tecnica che abbiamo sviluppato è semplice, versatile e, soprattutto, può essere svolta da qualsiasi laboratorio di biologia cellulare o molecolare”, afferma Anthony KL Leung, Professore associato presso la Bloomberg School nel dipartimento. di Biochimica e Biologia Molecolare. “La nostra tecnica consente anche di risparmiare tempo – da una a due ore contro più di due giorni utilizzando la tecnica precedente”.
Vedi anche, Nuovi approfondimenti sui tumori aggressivi guidati da RAS.
La ribosilazione dell’ADP è un processo che comporta l’attacco di una o più molecole di ribosio ADP a una proteina.
È tra le varie modificazioni chimiche reversibili che le cellule comunemente usano per mettere a punto le attività di diverse proteine ed è nota per supportare una varietà di importanti processi cellulari, inclusa la riparazione del DNA danneggiato.
Gli inibitori di PARP sono progettati per colpire tumori che hanno già difetti esistenti nelle riparazioni del DNA. Di conseguenza, questi tumori si basano sulle vie di riparazione del DNA residuo mediate da PARP per la sopravvivenza. Mediante l’inibizione di queste vie residue mediate da PARP, le cellule tumorali possono essere uccise, risparmiando allo stesso tempo cellule sane con riparazione funzionale del DNA. I farmaci inibitori di PARP sono ora usati per trattare i pazienti con i geni “ereditari” BRCA nei tumori ovarici, mammari e di altro tipo.
Gli scienziati negli ultimi anni hanno trovato prove che suggeriscono che l’eccesso di ribosilazione di ADP contribuisce anche al morbo di Parkinson, alla malattia di Alzheimer e ad altre forme di neurodegenerazione. Uno studio dei laboratori di Leung e colleghi nel 2017 ha trovato prove che alcuni virus pericolosi annullano la ribosilazione di ADP per facilitare l’infezione.
In precedenza, i ricercatori avevano solo tecniche limitate e relativamente ingombranti per studiare la ribosilazione dell’ADP in laboratorio. In particolare, il metodo tradizionale, basato sulla chimica organica, per attaccare i beacon molecolari o le maniglie chimiche ad ADP-ribosio richiede un setup complesso, impiega giorni per realizzarsi, funziona con bassa efficienza e funziona solo su molecole di ADP-ribosio “libere” al contrario ad ADP-ribosio legato a uno dei suoi bersagli proteici.
Nel nuovo studio, Leung e colleghi hanno dimostrato che potrebbero utilizzare un enzima chiamato oligoadenilato sintetasi 1 per attaccare in modo efficiente una molecola chiamata dATP a un’estremità di ADP-ribosio, sia libera che legata alle proteine. Hanno dimostrato che potevano attaccare non solo semplici dATP, ma anche varianti o analoghi di dATP che incorporano atomi fluorescenti o radioattivi che sono utili, ad esempio, nello studio di come la ribosilazione dell’ADP influisce sulle funzioni delle proteine che modifica.
Gli scienziati hanno anche scoperto che potevano attaccare analoghi dATP che funzionano come maniglie chimiche, consentendo l’isolamento anche di tracce di proteine ADP-ribosilate da una soluzione al fine di identificare le proteine che sono state modificate in questo modo.
Le molecole di ADP-ribosio si attaccano spesso alle proteine non singolarmente, ma in unità o polimeri simili a catene noti come poli-ribosio ADP (PAR). Leung e colleghi hanno dimostrato che la loro nuova tecnica può essere utilizzata per misurare le lunghezze di questi polimeri PAR sulle proteine a cui sono attaccati. Il nuovo metodo consente quindi lo studio del modo in cui la lunghezza del polimero PAR determina l’effetto della ribosilazione dell’ADP su una data proteina. In linea di principio, lo stesso approccio potrebbe essere usato per valutare quanto bene gli inibitori di PARP riducano la lunghezza del polimero PAR su proteine chiave e in che modo tale riduzione di lunghezza sia correlata con gli esiti del cancro.
Gli scienziati hanno chiamato la loro nuova tecnica, “ELTA” – Etichettatura enzimatica del terminale ADP-ribosio -.
“Si può pensare a ELTA come ad un adattatore per le attuali tecniche di biologia molecolare per studiare il polimero PAR“, afferma Leung.
Leung ed i suoi colleghi si aspettano che ELTA sarà ampiamente utilizzata nello studio della ribosilazione dell’ADP e dei processi correlati, sia per la biologia pura che per la ricerca rivolta alle applicazioni cliniche. “Potrebbe consentire nuovi metodi diagnostici che utilizzerebbero la lunghezza del polimero PAR come biomarker per cancri e altre malattie”, afferma Leung.
Fonte, Johns Hopkins