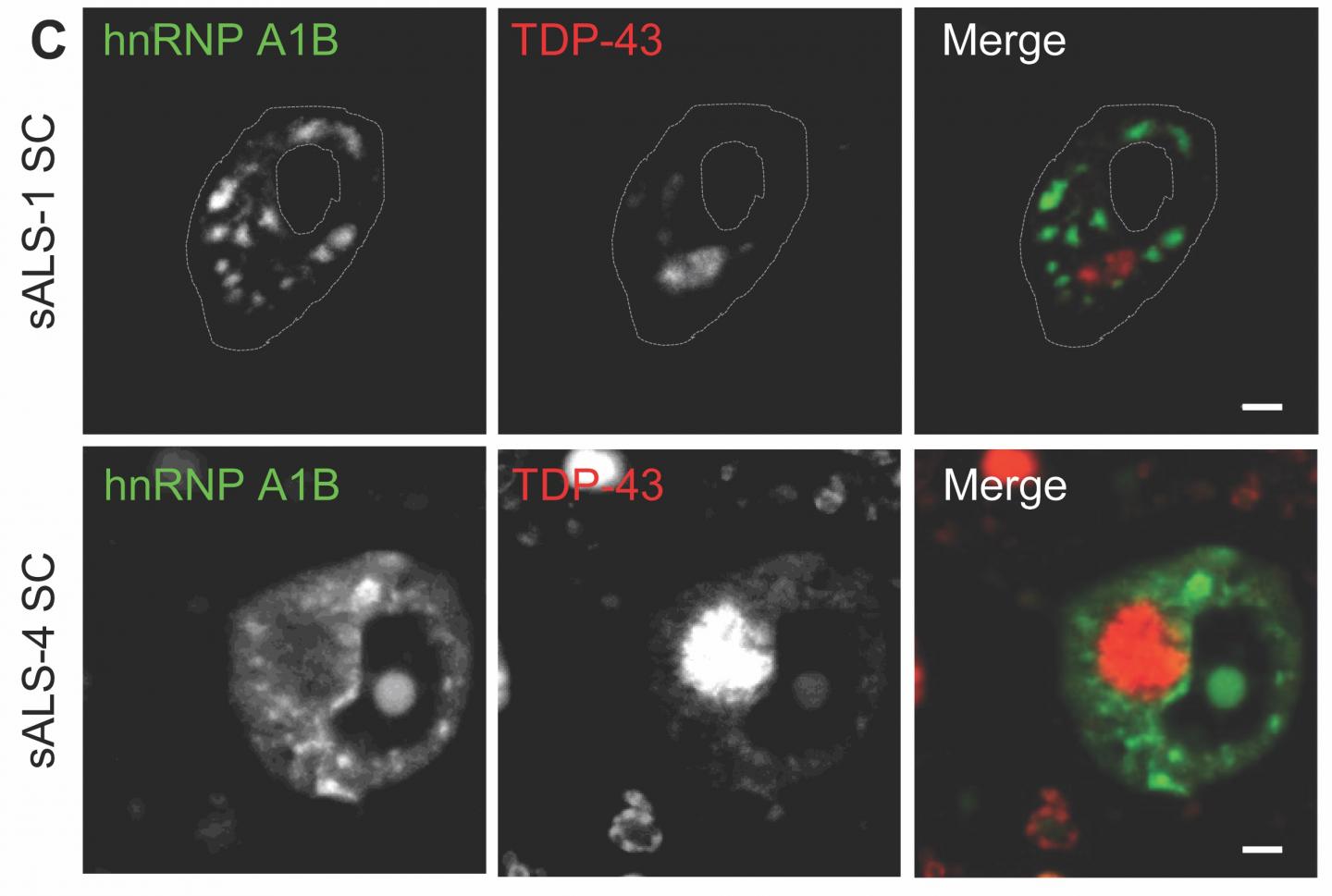
Immagine: hnRNP A1B è espresso nel SNC e forma aggregati citoplasmatici nei neuroni del paziente SLA. Credit: CRCHUM.
Ci sono voluti otto lunghi anni di ricerca, ma ora un team internazionale guidato da neuroscienziati all’Université de Montréal ha scoperto un meccanismo molecolare di base che aiuta a capire meglio come funziona la malattia di Lou Gehrig o sclerosi laterale amiotrofica (SLA).
E quella scienza di base potrebbe un giorno portare a una nuova terapia per questa malattia debilitante, che paralizza le persone rimuovendo la capacità del cervello di comunicare con i loro muscoli, portando infine alla paralisi e alla morte prematura.
“È una storia di ricerca fondamentale su ciò che accade normalmente nelle cellule del corpo e sui cambiamenti nel contesto della SLA”, ha detto Jade-Emmanuelle Deshaies, ricercatrice associata in neuroscienze presso il Centro di ricerca dell’Ospedale di UdeM (CRCHUM) e autrice principale del studio congiunto Canada-Israele, pubblicato online oggi sulla rivista Brain.
La ricerca è iniziata otto anni fa quando Deshaies e il suo supervisore, Professore associato di neuroscienze Christine Vande Velde, hanno iniziato a indagare su ciò che accade a varie molecole quando TDP-43, una proteina che lega i “messaggeri” nella cellula conosciuta collettivamente come RNA e che è centrale per patologia SLA, viene rimossa dal nucleo.
“Il nostro focus specifico era su altri tipi di proteine leganti l’RNA che potrebbero essere attori interessanti nella malattia”, ha ricordato Deshaies. “Uno di questi, hnRNP A1, ha attirato la nostra attenzione, in particolare perché c’era una seconda forma che è raramente menzionata in letteratura”.
Ma prima, un po ‘di scienza di base.
Nella biologia molecolare, i geni codificano l’RNA che viene quindi tradotto in proteine, i cavalli da lavoro delle cellule. Esistono molte versioni differenti di RNA, ognuna codificante per diverse versioni di una proteina. TDP-43, per esempio, lega l’RNA e può cambiare il modo in cui è stato unito – in una sequenza di ABCD, per esempio, o di ABCEFG – un processo chiamato splicing alternativo. Un’altra proteina legante l’RNA è hnRNP A1, nota in due varianti, entrambe regolate da TDP-43.
( Vedi anche:SLA: migliore comprensione di come i motoneuroni rispondono alla malattia).
Perché questo è importante per capire la SLA? Perchè TDP-43 è noto per essere un componente importante delle sostanze non viventi nella cellula chiamate inclusioni citoplasmatiche, che sono osservate nel 97% dei casi di SLA.
“I dati che abbiamo mostrano che quando TDP-43 è assente dal nucleo, è possibile modificare il modello di splicing di hnRNP A1“, ha affermato Vande Velde. “Il quadro generale è che esiste uno spettro molto più ampio di errata regolazione del metabolismo dell’RNA rispetto a quanto si pensava in precedenza e con ciò otteniamo una maggiore comprensione di ciò che sta andando male nella SLA e, data questa nuova conoscenza, possiamo potenzialmente sviluppare una terapia che prende di mira questo meccanismo “.
Uno sviluppo parallelo è la ricerca su un’ altra malattia dei motoneuroni, l’atrofia muscolare spinale (SMA). Gli scienziati sanno che l’hnRNP A1 gioca un ruolo nel suo progresso, controllando lo splicing di un importante gene chiamato SMN, il neurone motore di sopravvivenza. Vande Velde e il suo team non sanno ancora se la nuova variante di giuntura che hanno scoperto modifichi i livelli o la funzione SMN, ma indicano una nuova terapia farmacologica annunciata lo scorso anno per la SMA che mira allo splicing di SMN da hnRNP A1.
“Il farmaco è Nusinersen, venduto commercialmente come Spinraza”, ha spiegato Vande Velde. “Quando viene somministrato ai bambini abbastanza presto dopo la diagnosi di SMA, puoi gestire la loro atrofia muscolare spinale. I bambini che non erano in grado di rotolarsi o camminare, dopo il trattamento possono farlo “.
Sviluppato dagli scienziati del Cold Spring Harbor Laboratory e Ionis Pharmaceuticals, Nusinersen è uno sviluppo entusiasmante “perché è una terapia che nasce dalla comprensione di un meccanismo molecolare che coinvolge lo splicing alternativo”.
“Ci sono voluti molti anni per arrivare a quel punto, e allo stesso modo, il nostro lavoro è solo il primo ingranaggio al volante”, ha aggiunto la ricercatrice. “Che ci sia o meno un’influenza sull’espressione o sullo splicing del gene SMN o di altri geni importanti per la sopravvivenza dei motoneuroni, è qualcosa che dobbiamo valutare”, ha detto Vande Velde.
Il lavoro del suo laboratorio è anche una storia di persistenza. Come dice Deshaies, “la scienza è raramente semplice, spesso prende una strada tortuosa prima di portare a spiegazioni e alla vera comprensione di ciò che osserviamo”.
“Stiamo studiando un meccanismo che non è mai stato segnalato prima“, ha osservato Vande Velde. “Abbiamo avuto alcuni risultati contrastanti nelle prime fasi e ci è voluto del tempo per capire quale fosse il ruolo diTDP-43 in tutto questo: è stata” una grande impresa di squadra “, ha aggiunto,” con i membri del team israeliani (presso l’Università ebraica ), Quebec (all’UdE e all’Université de Sherbrooke) e altrove in Canada (all’università occidentale), tutti hanno dato un contributo fondamentale “.
Più ricerche della SLA vengono condotte in Quebec rispetto a qualsiasi altra provincia e lo studio guidato da UdeM è stato sostenuto da sovvenzioni da NSERC e dalla società non-profit SLA Society of Canada.
“Penso che sia un servizio importante comunicare ai pazienti e alle loro famiglie quali scoperte sono state fatte con le loro donazioni, come quelle ricevute attraverso la Ice Bucket Challenge”, ha concluso Vande Velde.
Fonte: Brain